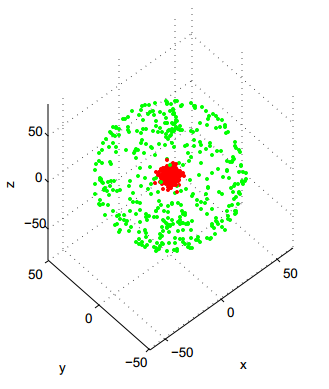
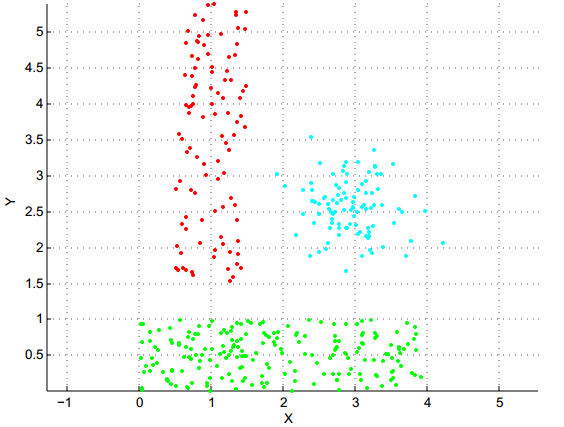
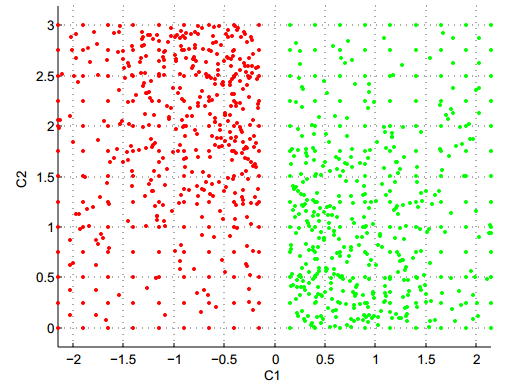
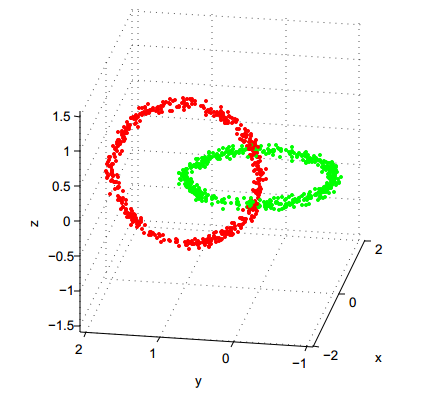
1. Comparaison des techniques classiques de clustering
- Se placer dans votre copie locale du répertoire des exemples :
- Charger dans R les 5 fichiers de données Atom, Lsun, WingNut, Chainlink, TwoDiamonds (visualisés ci-dessus) :
- tmp = read.table("xxx.lrn", comment.char = "%") #
où xxx doit valoir successivement Atom Lsun WingNut Chainlink et
TwoDiamonds
- true_data_xxx =tmp[,-1]
- Essayer sur ces bases d'exemples l'algorithme k-means :
- km_xxx = kmeans(true_data_xxx,K)
# où K est le nombre (à choisir par vous !) de
clusters à utiliser pour partitionner les données
- plot(true_data_xxx,col=km_xxx$cluster)
Donne-t'il toujours le résultat espéré ?
En analysant/comparant les formes des clusters des bases sur lesquelle
k-means fonctionne le mieux et le moins bien, essayer de comprendre
pour quels types de distributions de points k-means est
inadapté.
Confirmer votre intuition en tentant, sur une des bases pour lesquels
k-means fonctionne mal, de relancer k-means en demandant un nombre de
clusters supérieur au nombre réel, et en examinant les
formes des clusters obtenus.
- Tester maintenant le clustering par aggrégation hiérarchique ascendante (fonction hclust) :
dist2_xxx=dist(true_data_xxx)
hs_xxx=hclust(dist2_xxx,method="single")
plot(hs_xxx) # Pour visualiser le dendrogramme
labS_xxx=cutree(hs_xxx,K)
# K : nb de clusters voulus, qui déterminera hauteur où
le dendrogramme sera coupé
plot(true_data_xxx,col=labS_xxx)
hc_xxx=hclust(dist2_xxx,method="complete")
plot(hc_xxx) # Pour visualiser le dendrogramme
labC_xxx=cutree(hc_xxx,K) # K : nb de clusters voulus, qui déterminera hauteur où le dendrogramme sera coupé
plot(true_data_xxx,col=labC_xxx)
Sur quelle(s) base(s) d'exemples le "single-linkage" donne-t'il un résultat excellent ? Pourquoi est-ce logique ?
Inversement, sur quelle base le "single-linkage" fonctionne-t'il le moins bien, et pourquoi est-ce attendu ?
En comparant avec ce que vous aviez constaté pour k-means,
quelle variante (entre single-linkage et complete-linkage) vous semble
la + complémentaire de k-means ?
- [Facultatif] Essayer enfin le clustering spectral (fonction specc, accessible après avoir fait "library(kernlab)") :
- library(kernlab)
- sc_xxx_K = specc(true_data_xxx, centers=K) # où K est le nombre voulu de clusters
- plot(true_data_xxx, col=sc_xxx_K)
- help(specc) # Pour avoir l'aide complète sur l'ensemble des paramètres possibles de la fonction